chapter02 创建数据集
创建数据集
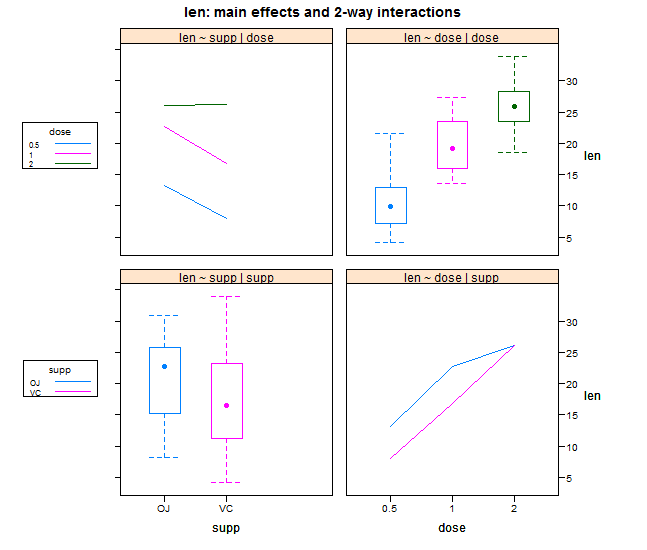
1. 向量
|
|
- 单个向量的数据类型必须一致
2. 标量
只含一个元素的向量:f<-3
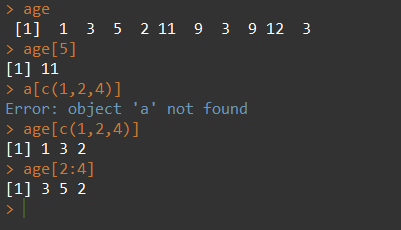
3. 向量的访问:
4. 矩阵
矩阵每个元素相同的模式

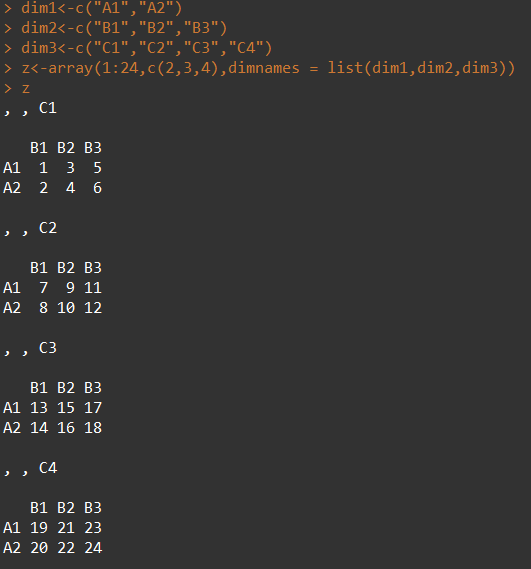
5. 数组:
6. 数据框
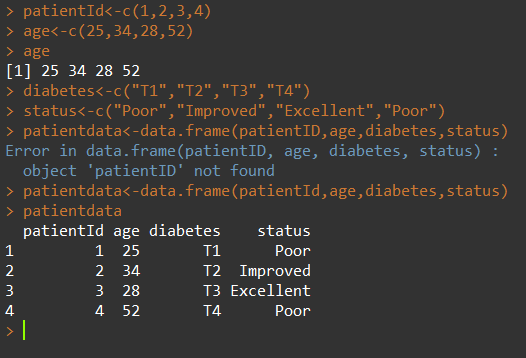
最常用的数据结构,可以包含不同的数据模式(数据类型)mydaya <- data.frame(col1,col2,col3),col1~col3可以为任何类型
数据框变量内容的读取:
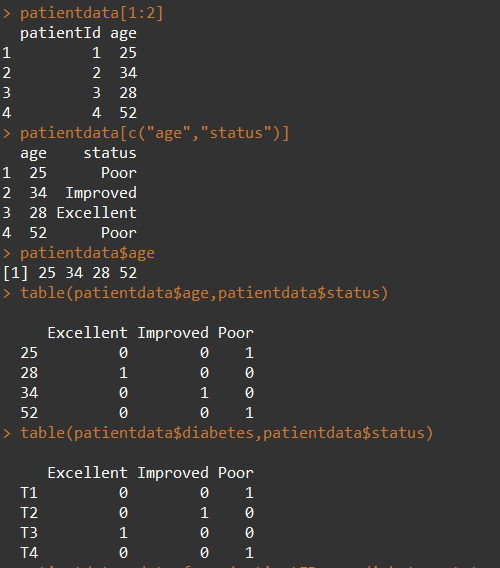
其中,变量名可以进行局部化:
|
|
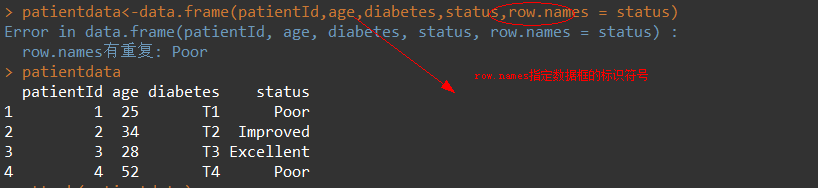
7. 因子
变量可归结为名义型、有序型或连续型变量,
- 名义型-无顺序(类别标识,男,女)-因子
- 有序型-有递进顺序,但不连续(病情的情况,好转,一般,差)-因子
- 连续型-有顺序的任意范围内值(年龄)
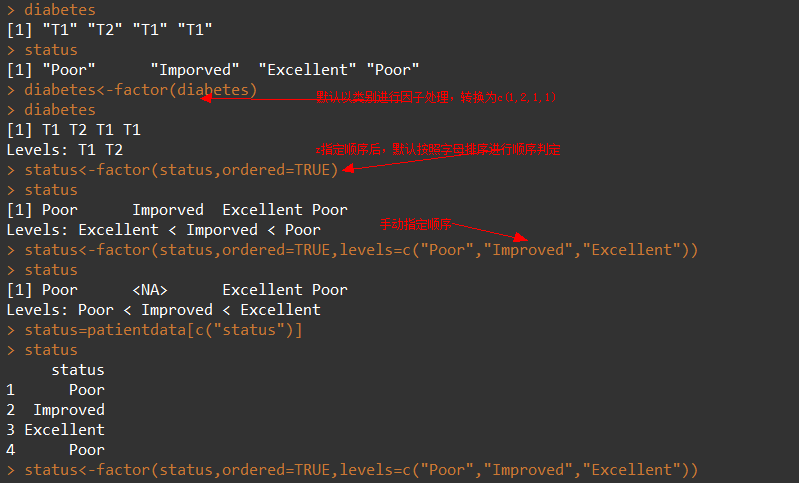
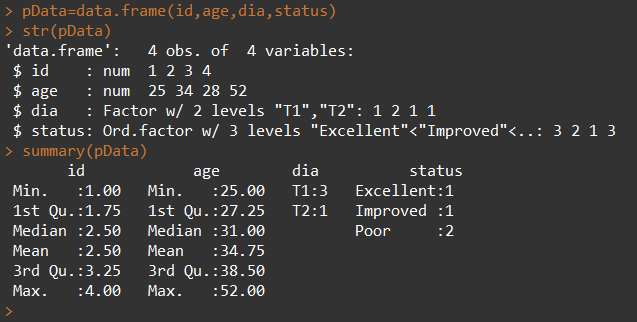
|
|
8. 列表
对象的有序集合,可以为任意对象:
|
|

9. 自动扩展特性
将一个值赋给某个向量、矩阵、数组或列表中一个不存在的元素时, R将自动扩展这个数据结构以容纳新值。

10. 删除变量
rm(var1)
11. 导入csv:
|
|
12. 变量标签与值标签


13. 实用函数
length(object)显示对象中元素/成分的数量dim(object)显示某个对象的维度str(object)显示某个对象的结构class(object)显示某个对象的类或类型mode(object)显示某个对象的模式names(object)显示某对象中各成分的名称c(objectt,…)将对象合并入一个向量cbind(object,…)按列合并对象rbind(object, …)按行合并对象Object输出某个对象head(object)列出某个对象的开始部分tail(object)列出某个对象的最后部分ls()显示当前的对象列表rm(object, …)删除一个或更多个对象。语句rm(list = ls())将删除当前工作环境中的几乎所有对象newobject <- edit(object)编辑对象并另存为newobject fix(object) 直接编辑对象